Le jeudi 28 novembre 2024, nous avons écouté deux présentations invitées
MANOCCA, une nouvelle approche statistique pour évaluer des effets sur la covariance/corrélation
Christophe Boetto, INRAE, MIA-Paris-Saclay
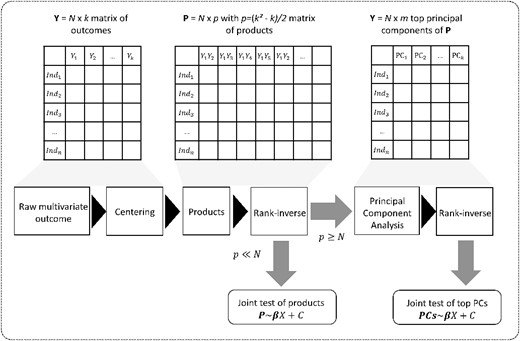
La présentation détaille la méthode statistique MANOCCA (Multivariate ANalysis of Conditional CovAriance) développé pendant ma thèse. Ce test évalue l’effet d’un prédicteur d’intérêt noté X (Age, Sexe, BMI, tabagisme, variant génétique, …) sur la covariance d’une variable dépendante multivariée noté Y (niveaux de métabolites, expression de gènes, espèce microbiennes, …) tout en ajustant pour des covariables C. Le test évalue donc le modèle Cov(Y) ~ X + C pour n’importe quel type de prédicteur binaire, catégoriel ou continue. Le travail a donné lieu à deux cas d’applications, le premier en microbiologie pour caractériser les changements d’intéractions de bactéries en fonction de facteurs environnementaux. Et le second dans la génétique du métabolisme pour évaluer des variants génétiques impactant la co-régulation de métabolites.
Reference :
Christophe Boetto, Arthur Frouin, Léo Henches, Antoine Auvergne, Yuka Suzuki, Etienne Patin, Marius Bredon, Alec Chiu, Milieu Interieur Consortium, Sriram Sankararaman, Noah Zaitlen, Sean P Kennedy, Lluis Quintana-Murci, Darragh Duffy, Harry Sokol, Hugues Aschard, MANOCCA: a robust and computationally efficient test of covariance in high-dimension multivariate omics data, Briefings in Bioinformatics, Volume 25, Issue 4, July 2024, bbae272, https://doi.org/10.1093/bib/bbae272
Available code in Python and R here
Create your own recipes steps for omics data: the {scimo} package,
Antoine Bichat, Laboratoires Servier

The rise of advanced high-throughput sequencing technologies has led to a massive increase in the production of omics data, encompassing genomics, transcriptomics, proteomics, metagenomics, and more. To effectively explore and analyze omics data, specialized preprocessing techniques are essential, including feature normalization, selection, and aggregation. However, many of these specific methods were not initially available in the original ‘recipes’ package. As a response, we have developed an extension package, {scimo}, designed to seamlessly integrate these techniques into the tidymodels ecosystem. {scimo} offers a comprehensive suite of preprocessing steps tailored for omics data analysis, while remaining adaptable to other data types. During this presentation, we will showcase the capabilities of {scimo} and provide insights into creating your own {recipes} extension package. Additionally, we will discuss strategies to navigate the potential pitfalls that we have encountered during the development process.
Informations pratiques
C’était un webinaire, une fois n’est pas coutume.